 Rによるデータ分析
Rによるデータ分析
 Rによるデータ分析
Rによるデータ分析
外れたサンプルの探索 をRで実施する時の例です。
このページは、特に外れたサンプルに注目したい時の方法になります。
高次元を2次元に圧縮して可視化 で外れたサンプルを探索する方法は、 Rによる高次元を2次元に圧縮して可視化 と手順が変わらないので、このページでは紹介していません。
外れたサンプルは、数が少ないので、通常のヒストグラムでは、見落とすことがあります。 ここでは、 ggplot2 に Plotly を組み合わせて、範囲を自由に変えられるグラフにしています。
library(ggplot2) # ライブラリを読み込み
library(plotly))# ライブラリを読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data1 <- read.csv("Data.csv", header=T) # データを読み込み
ggplotly(ggplot(Data1, aes(x=X1)) + geom_histogram()))# ヒストグラムを描く
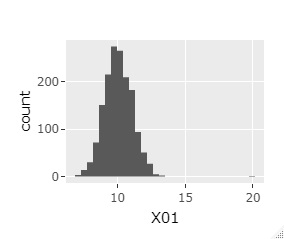
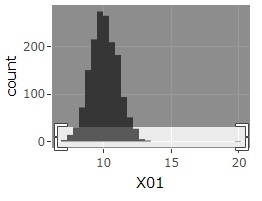
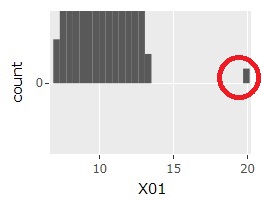
ヒストグラムは、最初、一番左のものができます。
真ん中のグラフのようにして、範囲を選択すると、一番右のものができます。
すると、20のあたりのサンプルが見えやすくになります。
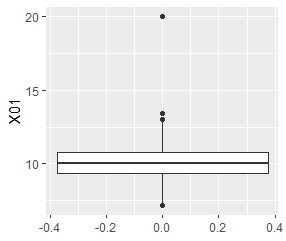
箱ひげ図 は、上下の四分位点の幅の1.5倍のところに、「ひげ」を描き、それよりも離れたサンプルは外れているものとして、「点」で表します。
library(ggplot2) # ライブラリを読み込み
library(plotly))# ライブラリを読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data1 <- read.csv("Data.csv", header=T) # データを読み込み
ggplot(Data1, aes(y=X1)) + geom_boxplot()# 箱ひげ図を描く
スミルノフ=グラブス検定は、1次元のデータについて、分布の一番外側のサンプルについて、外れ値がどうかを判定します。
library(outliers)# ライブラリを読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data <- read.csv("Data.csv", header=T) # データを読み込み
grubbs.test(Data[,1])# 1列目のデータをスミルノフ=グラブス検定

この場合は、最大値の「20」が外れ値かどうかを検定して、p値がかなり小さいので、外れ値と判定したことになります。
p値で判定することになるのですが、p値はサンプル数が多いと、小さな値になりやすいです。 筆者は目安として、「サンプル数が30位までの時は、p値が0.01よりも大きかったら、外れ値ではない」、と考えています。 30よりも大きいと、この方法で外れ値を定量的に判断はできないと思っています。
クラスター分析 の階層型を使う方法です。 階層型はいろいろありますが、外れ値を見つけやすいのは、最近接法(single)なので、下記はsingleにしています。
また、デンドログラムは、plotlyと組み合わせて、拡大できるようにしています。
library(plotly)
library(ggdendro)
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data1 <- read.csv("Data.csv", header=T) # データを読み込み
Data10 <- Data1[,1:2]## クラスター分析に使うデータを指定。ここでは1〜2列目の場合
Data11_dist <- dist(Data10)# サンプル間の距離を計算
hc <- hclust(Data11_dist, "single") # 最近接法による階層型クラスター分析
ggplotly(ggdendrogram(hc, segments = TRUE, labels = TRUE, leaf_labels = TRUE, rotate = FALSE, theme_dendro = TRUE))# 拡大可能なデンドログラム
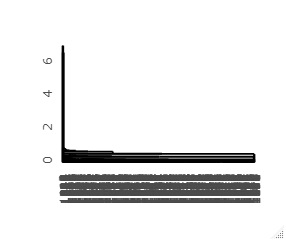
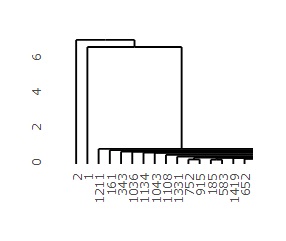
最初にできるデンドログラムが左で、そのデンドログラムの左側を拡大すると、右になります。
グラフの縦軸は、距離に相当するものなので、
1番目と、2番目のサンプルが、他の大多数からは離れていることが、このグラフからわかります。
output <- cutree(hc,k=3)# 3個にグループ分け
Data <- cbind(Data1, output) # 最初のデータセットにグループ分けの結果を付ける
Data$output <-factor(Data$output) # グループの変数を文字列型にする
ggplot(Data, aes(x=X1, y=X2)) + geom_point(aes(colour=output))# 二次元散布図 色分け・形分け
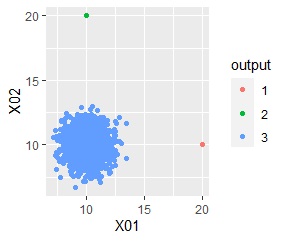
外れたサンプルは、「1」、「2」というグループになり、大多数が含まれれる「3」のグループと区別できています。
クラスター分析 のDBSCANを使う方法です。
library(ggplot2) # ライブラリを読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data1 <- read.csv("Data.csv", header=T) # データを読み込み
Data10 <- Data1[,1:2]## クラスター分析に使うデータを指定。ここでは1〜2列目の場合
Data11 <- Data10 # 出力先の行列を作る
for (i in 1:ncol(Data10)) { # ループの始まり。データの列数を数えて同じ回数繰り返す
Data11[,i] <- (Data10[,i] - min(Data10[,i]))/(max(Data10[,i]) - min(Data10[,i]))
} # ループの終わり
Data11_dist <- dist(Data10)# サンプル間の距離を計算
library(dbscan) # ライブラリを読み込み
dbs <- dbscan(Data11, eps = 0.2) # DBSCANで分類。epsは、コア点からの距離の範囲です。ここでは、0.2にしました。
output <- dbs$cluster # 分類結果の抽出
Data <- cbind(Data1, output) # 最初のデータセットにグループ分けの結果を付ける
Data$output <-factor(Data$output) # グループの変数を文字列型にする
ggplot(Data, aes(x=X1, y=X2)) + geom_point(aes(colour=output))# 二次元散布図 色分け・形分け
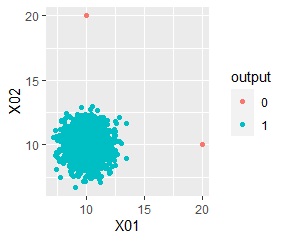
外れたサンプルは、「0」というグループになります。
LOF を使う方法です。
library(Rlof)# ライブラリを読み込み
library(ggplot2) # ライブラリを読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data <- read.csv("Data.csv", header=T) # データを読み込み
Data1 <- Data # 加工用のデータの作成
Data1 <- Data[,1:2]# 分析するデータを1から2列目で指定
LOF <- lof(Data1,5)# LOF。近傍の5個を使う場合
Data2 <- as.data.frame(cbind(LOF ,Data))# データを合体
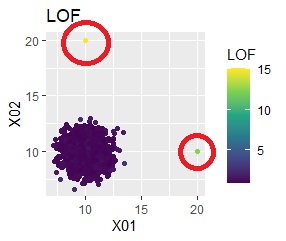
ggplot(Data2, aes(x=X1, y=X2)) + geom_point(aes(colour=LOF))+ggtitle("LOF")+ scale_color_viridis_c(option = "D")# 散布図を描く
ggplot(Data2, aes(y=LOF)) + geom_boxplot()# 箱ひげ図を描く
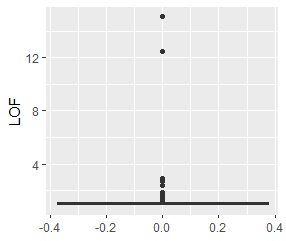
外れている2個のサンプルのLOFは、高く計算されました。
One-Class SVMで、分布の内側と外側を分ける方法です。
library(ggplot2) # ライブラリを読み込み
library(kernlab) # ライブラリを読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data1 <- read.csv("Data.csv", header=T) # データを読み込み
Data10 <- Data1[,1:2]# クラスター分析に使うデータを指定。ここでは1〜2列目の場合
Data11 <- transform(Data10, type=1)# ラベルの列を追加
OC <- ksvm(type~.,data=Data11,type='one-svc', kernel="besseldot", nu = 0.01)# nuは外れ値の割合
output <- predict(OC,Data11)# 分類結果の抽出
#カーネルがanovadot
OC <- ksvm(type~.,data=Data11,type='one-svc', kernel="anovadot", nu = 0.01)# nuは外れ値の割合
#カーネルがrbfdot
OC <- ksvm(type~.,data=Data11,type='one-svc', kernel="rbfdot", nu = 0.01)# nuは外れ値の割合
Data <- cbind(Data1, output) # 最初のデータセットにグループ分けの結果を付ける
Data$output <-factor(Data$output) # グループの変数を文字列型にする
ggplot(Data, aes(x=X1, y=X2)) + geom_point(aes(colour=output))# 二次元散布図 色分け・形分け
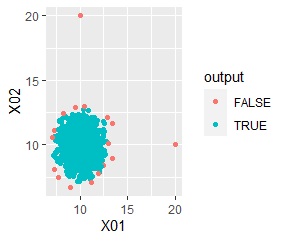
外れたサンプルは、「FALSE」というグループになります。

