Rによるデータ分析
Rによるデータ分析
 Rによるデータ分析
Rによるデータ分析
ベクトル量子化回帰分析 のRによる実施例です。
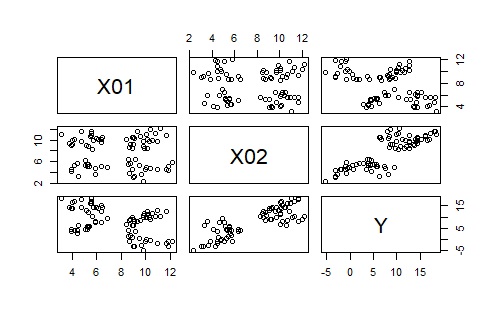
この実施例のデータは、以下のようなものです。
library(MASS)
library(mclust)
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data <- read.csv("Data.csv", header=T) # データを読み込み
Data10 <- Data
Data10$Y <- NULL# Yの列を削除
Data11 <- Data10 # 出力先の行列を作る
for (i in 1:ncol(Data10)) {
Data11[,i] <- (Data10[,i] - min(Data10[,i]))/(max(Data10[,i]) - min(Data10[,i]))# 正規化
}
mc <- Mclust(Data11,4) # 混合分布法で分類。これは4個のグループ分けの場合
cluster <- mc$classification # 分類結果の抽出
max_output <- max(cluster)
nrow_Data <- nrow(Data11)
dummy_Data <- data.frame(matrix(0, nrow_Data, max_output))
for (i in 1:nrow_Data) {
dummy_Data[i,cluster[i]] <- 1
}
Data5 <-cbind(Data,cluster)# データセットの作成
Data4 <-cbind(Data,dummy_Data)# データセットの作成
gm <- step(glm(Y~.^2, data=Data4, family= gaussian(link = "identity"))) # 2次の重回帰分析
summary(gm) # 結果の出力
#ここから予測の手順
library(ggplot2)
predicted_Y <- predict(gm,Data4)
Data4s2 <- cbind(Data4,predicted_Y)
ggplot(Data4s2, aes(x=Y, y=predicted_Y)) + geom_point()
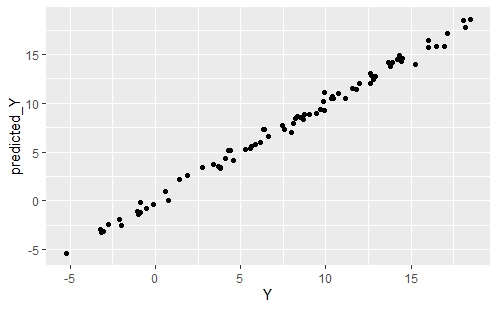
ほぼ一直線に並ぶので、非常に精度が高く予測できていることが、わかります。
ggplot(Data4s2, aes(x=X01, y=X02)) + geom_point(aes(colour=predicted_Y)) + scale_color_viridis_c(option = "D")
左が元のデータ、右が予測値です。
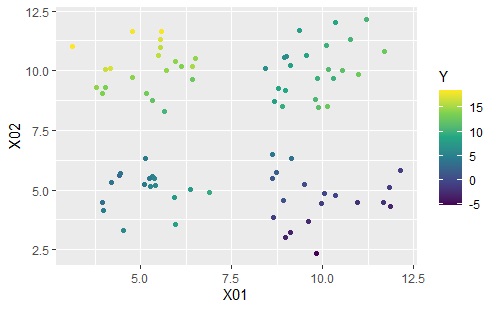
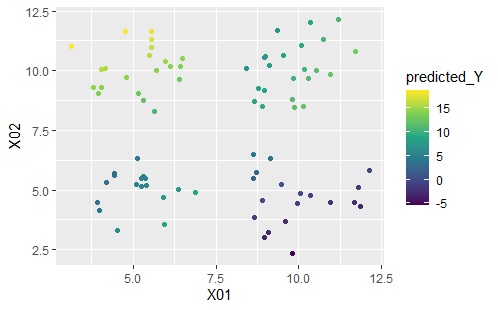
上記は、用意したサンプル全部についてモデルを作って、用意したサンプルと答え合わせをしています。
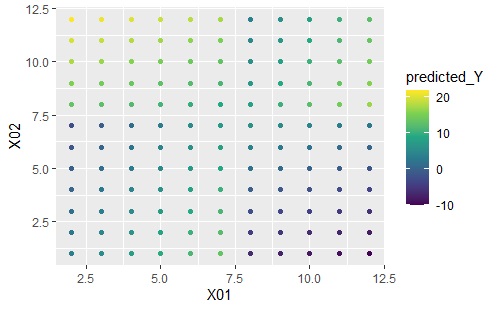
任意の位置について、予測値を求めます。 以下の例は、Data2.csvというファイルに、X01、X02という変数があり、データが書かれている場合です。
Data2 <- read.csv("Data2.csv", header=T) # データを読み込み
Data21 <- Data2
for (i in 1:ncol(Data2)) {
Data21[,i] <- (Data2[,i] - min(Data10[,i]))/(max(Data10[,i]) - min(Data10[,i]))
}
cluster <- predict(mc, Data21)$classification
nrow_Data2 <- nrow(Data2)
dummy_Data2 <- data.frame(matrix(0, nrow_Data2, max_output))
for (i in 1:nrow_Data2) {
dummy_Data2[i,cluster[i]] <- 1
}
Data52 <-cbind(Data2,cluster)# データセットの作成
Data42 <-cbind(Data2,dummy_Data2)# データセットの作成
predicted_Y <- predict(gm,Data42,type="response")
Data2s22 <- cbind(Data52,predicted_Y)
ggplot(Data2s22, aes(x=X01, y=X02)) + geom_point(aes(colour=predicted_Y)) + scale_color_viridis_c(option = "D")