Rによるデータ分析
Rによるデータ分析
 Rによるデータ分析
Rによるデータ分析
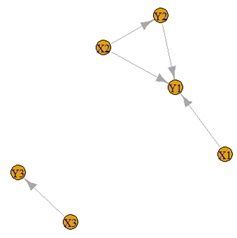
有向情報量分析 のRによる実施例です。
有向情報量分析の前処理として、 相互情報量行列 を計算します。
基本的に質的変数を扱う方法ですが、量的変数は 1次元クラスタリング の方法で、質的変数に変換するコードが入っているので、 質的・量的が混合していたり、量的変数だけでも使えるようにしてあります。
library(igraph) #ライブラリを読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data <- read.csv("Data.csv", header=T) # データを読み込み
Data1 <- Data # 1次元クラスタリングの出力先を作る
n <- ncol(Data1)
for (i in 1:n) {
if (class(Data1[,i]) == "numeric") { # 条件分岐の始まり
Data1[,i] <- droplevels(cut(Data1[,i], breaks = 3,include.lowest = TRUE))# 3分割する場合。量的データは、質的データに変換する。
}
}
GM2 <- matrix(0,nrow=n,ncol=n)
rownames(GM2)<-colnames(Data1) # 行名をつける
colnames(GM2)<-colnames(Data1) # 列名をつける
for (i in 1:n) {
for (j in 1:n) {
if( i >= j){
TableXY <- table(Data1[,i], Data1[,j])# 分割表の作成
Px <- TableXY
for (k in 1:nrow(TableXY)) {
Px[k,] <- sum(TableXY[k,])/sum(TableXY)
}
Py <- TableXY
for (k in 1:ncol(TableXY)) {
Py[,k] <- sum(TableXY[,k])/sum(TableXY)
}
Pxy <- TableXY/sum(TableXY)
pMI <- Pxy*log(Pxy/Px/Py,2)
pMI[is.na(pMI)] <- 0
MI <- sum(pMI)
k <- min(ncol(TableXY), nrow(TableXY))
MI <- sum(pMI)
GM2[i,j] <- MI# 相互情報量の書き込み
GM2[j,i] <- MI# 相互情報量の書き込み
}
}
}
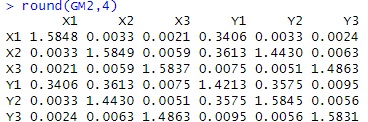
round(GM2,4)
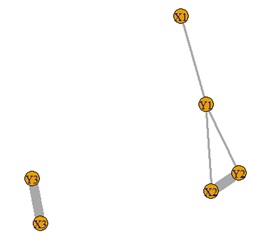
相互情報量行列を無向グラフにします。
この後の有向情報量分析では、相互情報量がしきい値よりも高く、かつ、平均情報量の差がしきい値よりも高くないと、 線が引かれませんが、ここでは相互情報量だけで線の有無が決まります。 そのため、有向情報量分析の、事前分析として役立ちます。
GM2[GM2<0.1] <- 0 # 係数の絶対値が0.1未満の場合は0にする(非表示にするため)
GM3 <- GM2
diag(GM3) <- 0
GM3 <- GM3*10 # 一番大きな値が10になるように修正(パスの太さを指定するため)
GM4 <- graph.adjacency(GM3,weighted=T, mode = "undirected") # グラフ用のデータを作成
plot(GM4, edge.width=E(GM4)$weight) # グラフを作成
directed_cor201 <- GM2
directed_cor201[,1:n] <- 0
for (i in 1:n) {
for (j in 1:n) {
if(i > j){
if(GM2[i,j] > 0.1){
diffGM2 <- GM2[i,i] - GM2[j,j]
if(abs(diffGM2) > 0.0001){
if(diffGM2 > 0){
directed_cor201[i,j] <- diffGM2
} else {
directed_cor201[j,i] <- -diffGM2
}
}
}
}
}
}
directed_cor201
GM5 <- ceiling(directed_cor201)
GM6 <- graph.adjacency(GM5,weighted=F, mode = "directed")
plot(GM6)