 Rによるデータ分析
Rによるデータ分析
 Rによるデータ分析
Rによるデータ分析
LiNGAM のRによる実施例です。
モデル式の係数を調べる方法です。
ここでは、以下の式で作ったデータを使っています。
eのところは、一様分布の乱数を使っています。
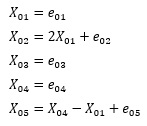
library(pcalg) #ライブラリの読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data <- read.csv("Data.csv", header=T) # データを読み込み
Str <- lingam(Data)$Bpruned # LiNGAM
rownames(Str) <- colnames(Data) # 行名を追記
colnames(Str) <- colnames(Data) # 列名を追記
Str
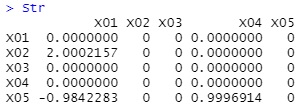
データを作った時の係数とほぼ同じ値が求まりました。
重回帰分析 で、各変数の影響の大きさを調べたい時は、単純に回帰式の各変数の係数を比べることは間違いで、偏回帰係数を比べます。 偏回帰係数は、各変数を 標準化 してから、重回帰分析をして求まる係数になります。
因果関係の分析にLiNGAMを使う場合も、単純にLiNGAMの出力を使おうとすると、同じ間違いが起きます。 ここでも標準化をしてから、LiNGAMを実行することにします。
そして、この係数を影響の大きさを表す数値として、ネットワーク構造の太さに使います。
#のついた3行は、#を消せば、データの前処理に標準化をします。 筆者の経験上、標準化をすると、矢印の方向が間違った結果になることが多いです。
library(pcalg) #ライブラリの読み込み
library(igraph) #ライブラリの読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data <- read.csv("Data.csv", header=T) # データを読み込み
#for (i in 1:ncol(Data)) {
# Data[,i] <- (Data[,i] - mean(Data[,i]))/sd(Data[,i]) #データを標準化
#}
Str <- lingam(Data)$Bpruned # LiNGAM
rownames(Str) <- colnames(Data) # 行名を追記
colnames(Str) <- colnames(Data) # 列名を追記
GM2 <- t(abs(Str)) # 絶対値にして転置する
GM3 <- GM2*5/max(GM2) # 値を適度な大きさにする
GM4 <- graph.adjacency(GM3,weighted=T, mode = "directed") # グラフ用のデータを作成
plot(GM4, edge.width=E(GM4)$weight) # グラフを作成
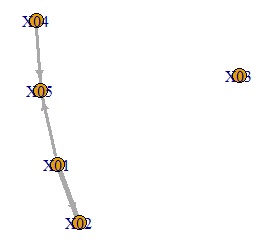
矢印の元側の変数に誤差が加わって、矢印の先の変数になっていることを表しているグラフができます。
参考文献のところにありますが、pcalgというライブラリを使うには、別のライブラリが先にインストールされている必要があります。 それが原因のようで、環境によっては、上記のコードはエラーになります。
そこで、pcalgを使わずにLiNGAMを実行するためのコードが下記になります。 筆者の自作のコードです。 R-EDA1 でも、このコードを使っています。
参考文献にあったハンガリアン法による行列の並び替えというのがわからなかったため、 対角成分の和が最小になる行列は、ランダムに行を並び変えたものを10000通り作って、その組合せの最小値を作るようにしています。 7の階乗が5040なので、 変数が8個以上になると、だいたい合っているものの、本当に最小の行列ではない可能性があります。
library(igraph) #ライブラリの読み込み
library(fastICA)
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data <- read.csv("Data.csv", header=T) # データを読み込み
n <- ncol(Data)
#for (i in 1:n) {
#Data[,i] <- (Data[,i] - mean(Data[,i]))/sd(Data[,i]) #データを標準化
#}
fICA <- fastICA(Data, n) #独立成分分析
K <- fICA$K
W <- fICA$W
tKW <- t(K %*% W)
rownames(tKW) <- colnames(Data)
colnames(tKW) <- colnames(Data)
MintKW <- tKW
MinSum <- sum(diag(1/abs(tKW)))
for (i in 1:10000) {
Randomized <- order(sample(tKW,n))
tKW2 <- tKW[Randomized,Randomized]
SumdiagRabstKW <- sum(diag(1/abs(tKW2)))
if(MinSum > SumdiagRabstKW){
MinSum <- SumdiagRabstKW
MintKW <- tKW2
}
}
MintKW2 <- MintKW
for (i in 1:n) {
MintKW2[i,] <- MintKW2[i,]/MintKW2[i,i] #対角成分が1になるように各行を割る
}
Str <- MintKW2
for (i in 1:n) {
for (j in 1:n) {
if(i >j){
if(abs(Str[i,j]) > abs(Str[j,i])){
Str[j,i] <- 0
} else {
Str[i,j] <- 0
}
}
}
}
diag(Str) <- 0 # 対角成分を0にする
GM2 <- t(abs(Str)) # 絶対値にして転置する
GM3 <- GM2*5/max(GM2) # 値を適度な大きさにする
GM3[GM3<3] <- 0 # 3未満の場合は0にする(非表示にするため)
GM4 <- graph.adjacency(GM3,weighted=T, mode = "directed") # グラフ用のデータを作成
plot(GM4, edge.width=E(GM4)$weight) # グラフを作成
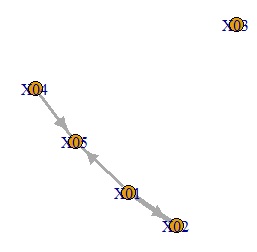
LiNGAMが有効な分布 のページに2つの方法の比較があります。
行列の分解 の一種に、「シュール分解」と呼ばれるものがあります。
シュール分解では、上三角行列が求まります。
LiNGAMでは、非巡回有向グラフを、三角行列で表せるところがポイントになっているので、 シュール分解を使うLiNGAMのアルゴリズムができるのではないかと思ったのですが、 筆者は、うまく行きませんでした。
ちなみに、Rでは、分解したい行列がAの時に、以下のようにして、上三角行列が求まります。
library(expm)
Schur(A)$T
筆者が試したところ、上三角行列に近い行列が求まるのですが、完ぺきな上三角行列にはなりませんでした。
「つくりながら学ぶ! Pythonによる因果分析 因果推論・因果探索の実践入門」 小川雄太郎 著 マイナビ出版 2020
LiNGAMの実装は、この本を参考にさせていただきました。
「「統計的因果探索」の一部を動かしてみた」
山川信之氏のブログです。
pcalgをインストールする前に、別のライブラリのインストールが必要なことが書かれています。
また、LiNGAMの簡単な実施例もあります。
https://techblog.nhn-techorus.com/archives/13805
の3つを紹介しています

