Rによるデータ分析
Rによるデータ分析
 Rによるデータ分析
Rによるデータ分析
高次元データのネットワーク分析 のRによる実施例です。
多次元データをスタートにする場合は、 高次元を2次元に圧縮して可視化 の方法として使えます。
この例では、Cドライブの「Rtest」というフォルダに、
「Data1.csv」という名前でデータが入っている事を想定しています。
library(igraph) #ライブラリを読み込み
setwd("C:/Rtest") # 作業用ディレクトリを変更
Data1 <- read.csv("Data1.csv", header=T) # データを読み込み
Data10 <- Data1[,1:3]# 分析する列を指定。この例は1から3列目の場合
Data11 <- (Data10 - apply(Data10,2,min))/(apply(Data10,2,max)-apply(Data10,2,min))# すべての変数のデータを、0から1の間のデータにする。
Data12 <- as.matrix(dist(Data11))# サンプル間の距離を計算して行列形式にする
Data12 <- (max(Data12) - Data12) # すべてのデータを符号を逆にする。
diag(Data12) <- 0 # 対角成分は0にする
Data12 <- Data12 / max(Data12) * 10# 0から10の間のデータにする。
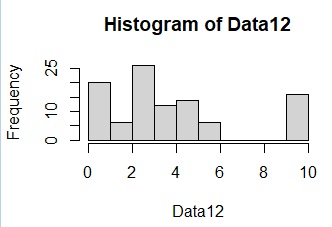
hist(Data12)# グラフにするデータをヒストグラムにする。
Data12[Data12< 8] <- 0 # 8未満の場合は0にする(非表示にするため)
GM4 <- graph.adjacency(Data12,weighted=T, mode = "undirected") # グラフ用のデータを作成
plot(GM4, edge.width=E(GM4)$weight) # グラフを作成
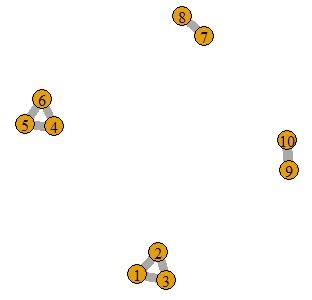
期待通りに4つのグループに分かれました。
多次元データをスタートにする場合のコードには、ヒストグラムでデータをチェックして、「8未満の場合は0」というコードが入っています。
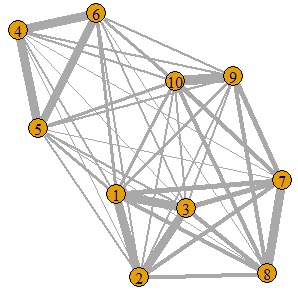
「8未満の場合は0」というコードを入れないと、下図のようになり、ゴチャゴチャして来ます。
もともとこの手法を使うときは、近いものを見つけたい時なので、遠いものとの線は切っておいても、問題はないかと思います。
なお、距離データをスタートにする場合でも、同じように遠いものとの線を切ることができます。